



Next: 1.4.5 分散関係線分データekdisp-line
Up: 1.4 入力データの設定法
Previous: 1.4.3 sample.ppf.sh
1.4.4 sample.atp.sh
デフォルトのファイルは、下記のようになっている。
シェル変数の部分は、上位のスクリプトで定義されている。
iatpflに1が入っている場合にのみ、このファイルは有効であり、
cpvo.shでiatpfl=0と指定している場合は、このファイルの編集は
行なわなくてよい。
cp ${srcdir}/atp.x ${rundir}
cp ${srcdir}/molatp.x ${rundir}
${rundir}/atp.x > ${rundir}/atp0 << end
${iatpfl}
${ibrav} ${cdm1} ${cdm2} ${cdm3} ${cdm4} ${cdm5} ${cdm6}
1 NSP
2 NA(IS)
0.0 0.0 0.0 TAU0(3,IA,IS)
0.25 0.25 0.25
0 IFMOL
0 IFSPG
end
if test ${ifmol0} = 0
then
exit
fi
cat > ${rundir}/molatp0 << end
2 NATM
1 0.0 0.0 -1.151690000000000E+00 ISP DTAU0(3,IA,IS)
1 0.0 0.0 1.151690000000000E+00
end
if test ${ifmol0} = 1
then
${rundir}/molatp.x > ${rundir}/atp1 << end
${rundir}/atp0
${rundir}/molatp0
1 NSP
2 NA(IS)
end
mv ${rundir}/atp0 ${rundir}/atp3
cp ${rundir}/atp1 ${rundir}/atp0
fi
exit
前半部分のIFMOLには常に、0を与える。
IFSPGは、0または1を与える。1のときは、ファイルspg0
から空間群に生成元を読み込んで、結晶の対称性を反映させた場所に原子が
置かれていないときにはその原子の座標と原子数を補って(20)に原子座標を出力し、
cpvoでその結果を使用する。
後半部分のifmol0=1に関係する部分は、分子を配置したい場合に用いる
ことができる。
使用する場合は、cpvo.sh中でifmol0=1と指定する。
atp.xの入力で指定した各座標に、分子を配置する。
上記の例では、1番目の原子種の原子で作られる2原子分子を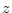 方向に向けて
配置する例である。合計で2分子、4原子の計算をすることになる。
入力座標
方向に向けて
配置する例である。合計で2分子、4原子の計算をすることになる。
入力座標DTAU0は、a.u.単位で入力する。中段のISPには、原子の種類
番号を指定する。現在は、同一方向に配向した分子を配置する場合だけに
対応している。
下記は、CaSiO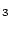 (ペロブスカイト型構造)の例である。
この場合
(ペロブスカイト型構造)の例である。
この場合ifmol=1に関係する部分は必要ないので、削除してある。
${rundir}/atp.x > ${rundir}/atp0 << end
${iatpfl}
${ibrav} ${cdm1} ${cdm2} ${cdm3} ${cdm4} ${cdm5} ${cdm6}
3 NSP
3 NA(IS)
0.0 0.5 0.5 TAU0(3,IA,IS)
0.5 0.0 0.5
0.5 0.5 0.0
1 NA(IS)
0.5 0.5 0.5 TAU0(3,IA,IS)
1 NA(IS)
0.0 0.0 0.0 TAU0(3,IA,IS)
end
exit
原子種が複数ある場合(NSPに2以上を指定する場合)は、
上記の例のように、
NA(IS), TAU0(3,IA,IS)をセットにして
データを作成する。




Next: 1.4.5 分散関係線分データekdisp-line
Up: 1.4 入力データの設定法
Previous: 1.4.3 sample.ppf.sh
Copyright (C), Tatsuki Oda (oda@cphys.s.kanazawa-u.ac.jp, Kanazawa University)